集成图结构和信息量的本体语义相似性度量
[关键词:集成图,信息量,本体语义] [热度 ]| 提示:此毕业设计论文完整版包含【论文,源程序,答辩稿】 作品编号:rjgc0366,word全文:38页,合计:14000字 |

本文将对关系特性相似性(Relative Specificity Similarity,RSS)的改进方法混合关系特性相似性(Hybrid Specificity Similarity,HRSS)进行介绍。HRSS是一种基于边以及基于信息量的语义相似性衡量方法,但是和RSS方法相比,HRSS方法并不完全依赖于基因本体的结构,这个方法还考虑到不同种群的注释量。本文还会对HRSS方法进行验证,并对其优缺点进行分析。
本体注释为在某些方面无法进行对比的本体提供了一种方法。例如,如果两个基因产物使用同一种方案进行注释的话,我们可以通过对比他们之间受到注释的条目来进行基因产物的比较。但是这种比较并不明显(例如,通过在一个交互的基因产物集合中寻找公共条目),因此有一种显式的比较,就是通过语义相似性来比较。语义相似性方法可以定义为,给出两个本体条目或者两个实体的注释条目集合,返回一个能够反映两者间关系的数值。
基因本体[1]在分子生物学中语义相似性的研究重点,不只因为它是生命科学理论中被最广泛采取的,同样也因为功能层面的基因产物比较对很多的应用来说都是十分关键的。基因产物中应用于GO注释到语义相似性为它们的功能相似性提供了一种方法。
研究内容和目标
本文的目的在于实现并验证RSS的改进算法HRSS在基因功能语义相似性比较的有效性。HRSS方法结合了基于IC值的节点,从而使得使用基于边的方法来测量语义相似性时不再只依赖于GO的结构。
本文的研究主要借助R语言的环境来进行,R语言中包含了许多与生物信息学、基因本体相关的包和数据库,通过R语言来测试和实现相对其他编程语言相对更容易实现,同时能够使效率得到有效提升。
在将HRSS算法借助R语言实现之后,我们将通过使用PPI数据库中的一些蛋白质相关条目进行验证,从而对HRSS算法进行评估。
论文结构安排
本文的剩余章节安排如下:
第二章对本文算法所涉及的领域以及背景知识进行介绍描述。首先对GO以及本体语义相似性的背景和发展进行介绍。然后介绍一些经典的语义相似性测量方法。而在这一章的最后部分会着重介绍RSS方法和改进于RSS方法的HRSS方法。
第三章主要描述了集成图结构和信息量的本体语义相似性度量中HRSS算法的具体实现方法。在这章中分为两个部分,第一部分主要对算法实现的环境与相应所需用到的资源进行介绍,第二部分会详细描述HRSS算法的实现步骤以及对该算法做出初步分析。
第四章将对本文中所实现的方法进行测试,然后对其结果进行详细分析,并与其他方法作相应的对比评估。
第五章对全文进行总结,对算法实现中碰到的问题进行描述,并提出相应解决方案,最后对下一步的研究工作进行了展望。
......
本文通过使用PPI数据集中的人类蛋白质基因,借助GO.db包,实现了基于GO的语义相似性度量方法HRSS。其中我们采用了两种常用的对比策略:MAX和BMA。一般来说BMA方法在评估直系同源的功能保守性和序列的相关性时与比MAX方法更好。而MAX策略由于只考虑到所有条目对最佳的匹配值,因此有个潜在的风险,就是可能会受到不正确的注释或者受到IEA注释[27]的干扰影响。这也许可以解释采用MAX策略时,测试结果并不够稳定。而MAX策略可以在两个基因产物共享一个相似功能但他们的相异度被低估时使用。因此,我们可以得出一个结论,MAX策略对两个基因产物的全局的功能相似性并不稳定,而BMA策略考虑到两个基因产物的所有条目对,但对于每个基因产物只挑出最佳匹配的相似值。总的来说,BMA优于MAX[10]策略。

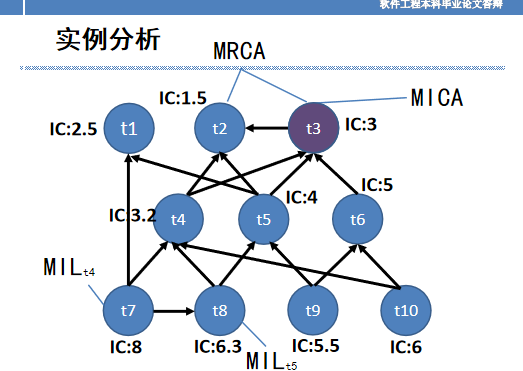
| 提示:此毕业设计论文完整版包含【论文,源程序,答辩稿】 作品编号:rjgc0366,word全文:38页,合计:14000字 |
本软件工程毕业设计论文作品由 毕业论文设计参考 [http://www.qflunwen.com] 征集整理——集成图结构和信息量的本体语义相似性度量(论文,源程序,答辩稿)!

 当前位置:
当前位置: 