基于组织特异数据的致病基因预测
[关键词:特异数据,致病基因] [热度 ]| 提示:此毕业设计论文完整版包含【论文,源程序,答辩稿】 作品编号:rjgc0383,word全文:48页,合计:17000字 |

本文整合了全局蛋白质相互作用网络和组织特异性信息,构建了组织特异的蛋白质相互作用网络,在此之上基于网络传播的思想设计并实现了基于组织特异的致病基因预测算法。实验结果表明,组织特异的蛋白质相互作用网络与之前的全局静态蛋白质相互作用网络相比,致病基因预测算法的预测准确性得到明显提高。
研究内容与目标
本文通过分析和研究组织特异基因的表达数据,设计并实现将组织特异性信息和现有的致病基因预测算法相结合的新的致病基因预测算法。
本文提出了两种具体的基于组织特异性的致病基因预测方法。这两种方案将组织特异性信息和已知的基于蛋白质相互作用网络的致病基因预测算法相结合。这两种方案的基本思路在于通过构建具有组织特异性的蛋白质相互作用网络,并将新构建的网络代替原有的蛋白质相互作用网络进行基因优先级排序,从而得出新的致病基因预测排名。
最后,本文使用留一交叉验证,对原有的致病基因预测算法和新构建的致病基因预测算法进行了比较和分析。
论文结构安排
本文剩余章节的内容安排如下:
第二章主要介绍致病基因预测算法和组织特异性的相关知识背景,具体包括致病基因预测算法所需的概念介绍和算法简介。
第三章主要介绍算法的原理和我们对算法的实现过程,具体包括PRINCE算法的实现和组织特异性算法的实现。
第四章主要介绍我们对算法的测试过程和测试结果,并对结果进行了相关分析。
第五章对全文进行总结,并提出了现有算法中仍然存在的一些问题,对进一步的研究工作进行了展望。
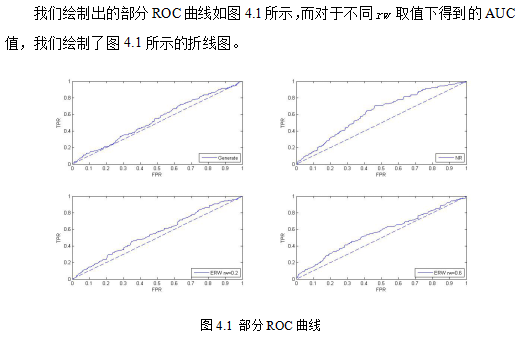
本文首先介绍了当前已有的基于蛋白质关联网络的致病基因预测算法,然后研究和实现了在蛋白质关联网络的基础上加入组织特异性信息后的致病基因预测算法。最后通过留一交叉验证和AUC等评估方式,讨论了基于组织特异性的致病基因预测算法的性能。
通过与原始通用网络的对比,组织特异性信息的加入增强了原始的致病基因预测算法的预测准确性。从不同角度对比节点移除法(NR方法)和边重赋权方法(ERW方法),我们认为宏观上看,ERW方法可以对更多的疾病预测出更好的致病基因,而从微观上看,NR方法针对某一特定疾病具有较高的致病基因预测准确率。因为ERW方法的预测准确率随着其参数的选择的变化而变化,因此,我们综合宏观和微观结果认为,ERW方法在时,从微观和宏观方面,具有较优的预测性能。
同时,本文也存在一些遗憾之处,主要在于我们没有能够充分利用基因的组织特异性表达数据,具体而言是我们没有能够将蛋白质相互作用网络中的基因与其中的基因完全匹配,这导致了预测结果的整体准确率较低。另外,我们没有能够对ERW方法的参数进行更为细致的测试与分析,我们不能排除ERW方法在取某一特定值时具有预测准确性高于NR方法的可能。

| 提示:此毕业设计论文完整版包含【论文,源程序,答辩稿】 作品编号:rjgc0383,word全文:48页,合计:17000字 |
本软件工程毕业设计论文作品由 毕业论文设计参考 [http://www.qflunwen.com] 征集整理——基于组织特异数据的致病基因预测(论文,源程序,答辩稿)!

 当前位置:
当前位置: 